Table of Contents
こんにちは、石橋です。
Facebookから面白い論文が出てきました。 以前、僕はalphafoldに関する記事を書きましたが、 最近は機械学習が生物学へのドメインに転用されて大きな精度向上をもたらしています。

このAlphaFoldではタンパク質の折り畳みに特化したモデルが提案されていましたが、今回はバイオインフォマティクス全体に利用できる可能性がある、生物版言語モデルをtransformerで構築したというものです。
Facebookではこんなことも研究されているんですね。
近年の自然言語処理界隈でよく行われる、大規模コーパスで事前学習し獲得した言語モデルを特定のタスクに応用するという流れを、タンパク質のドメインに持ち込み調査した興味深い研究です
この記事内の画像や表は論文からの引用です。
アブスト
- 教師なし学習を使用して、多様な2億5000万個のタンパク質に含まれる860億個のアミノ酸を対象とした深層文脈的言語モデルを学習
- 学習された表現空間は、生化学的性質のレベルから構造を反映したマルチスケールの組織を持っている
- 表現学習は、様々な用途に一般化する特徴を生み出し、長いレンジのコンタクト予測にも適応できる
TL;DR
- 自然言語に有効なモデルや目的関数が、ドメイン間の違いを超えて伝達されるかどうかは不明
これを受けて、この論文では
- 教師なしの表現に、生物学的な組織化原理や生物学的特性に関する情報が存在するかどうかをこの研究では調べた
その結果
- 表現空間には、物理化学的なスケールから遠隔地の相同性まで、組織化原理に合致した計量構造が見出され
- タンパク質の二次構造や三次構造が表現の中で識別できることもわかった。
- フォールディングに限らず、様々なタスクに応用できるように一般化された
とあります。 この研究のコントリビューションとしてまとめられていたポイントは以下の通りです
配列データから生物学的特性を学習することは、生物学のための生成的・予測的な人工知能を実現するための論理的なステップである。ここでは、進化の多様性にまたがる配列に対して、教師なし学習による深層文脈言語モデルをスケーリングすることを提案する。その結果、事前学習により獲得した表現の中に、二次構造、接触、生物学的活性などのタンパク質の基本的な特性に関する情報が現れることがわかった。学習した表現は、リモートホモロジーの検出、二次構造の予測、長距離の残基と残基の接触、突然変異の影響などのベンチマークに有効であることを示している。教師なしの表現学習は、変異効果と二次構造の最先端の教師付き予測を可能にし、長距離接触の予測のための最先端の特徴を改善する。
なるほど。 この表現学習をすることで、バイオインフォの分野の様々なタスクに応用ができるということですね。
2億5000万種類の多様なタンパク質配列で言語モデルの拡張
この研究では、UniParc databaseという2億5000万のタンパク質のデータベースを利用しています。 860億個のアミノ酸が含まれており、これは昨今の自然言語処理における大容量のニューラルネットワークアーキテクチャの学習に使用されている大規模テキストデータセットに匹敵するサイズです。
また、学習のベースラインとして表現学習と生成モデルにおいて実績を多く出しているtransformerを用いた。事前学習はMLMというタスクを採用。これはBERTと同じですね。
直感的なイメージとしては、マスクされた位置の予測を行うためには、モデルは、マスクされた部位と配列のマスクされていない部分との間の依存関係を特定しなければならないため、学習後には表現獲得ができていると考えられるということです。
Evaluation of Language Models
最初はtransformer以外のモデルも含めて比較をしています。 その結果が表1にまとめられています。
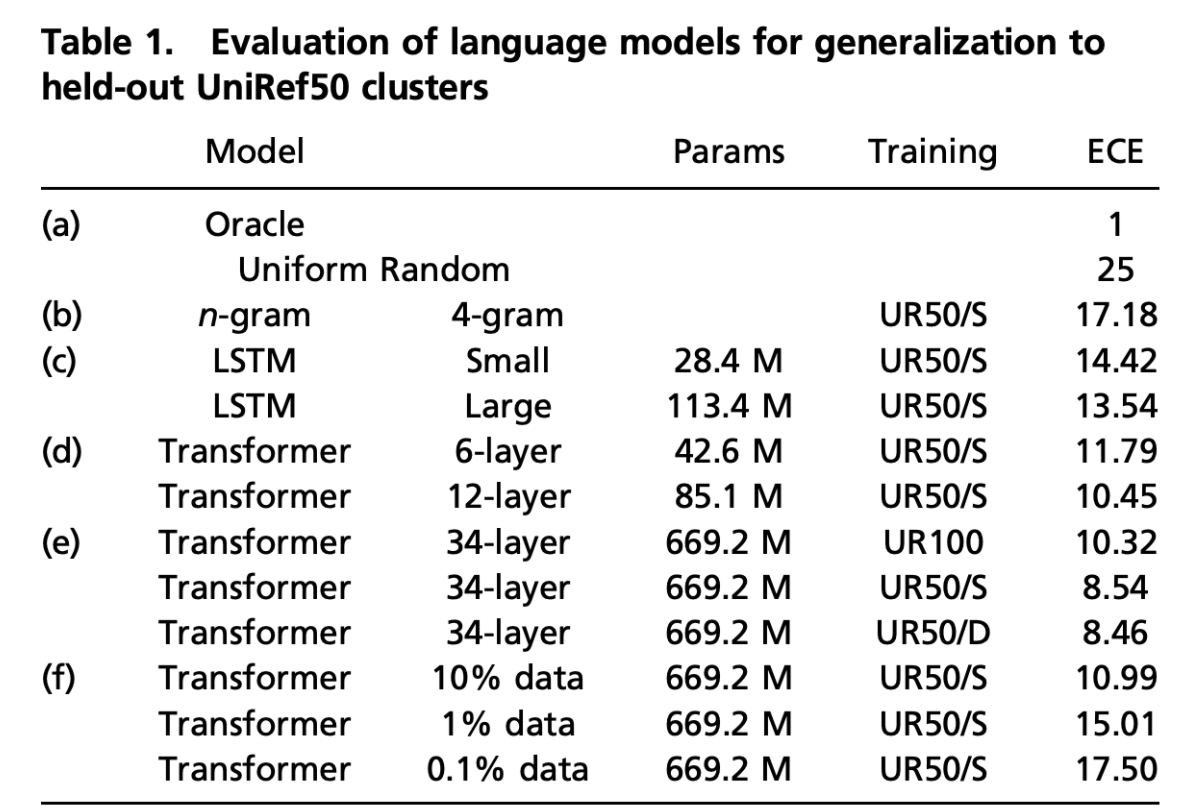
この表の、(c)と(d)は LSTMとtransformerの比較をしており データセットは同じで、モデルとパラメータが違います。
この比較から、transformerの方が少ないパラメータ数でlossが小さいことが見て取れますね。
また、(d)と(e)のUR50/Sに注目して比較してみると、モデルのパラメータ数が多いほどlossが小さいことがわかります。つまり、モデルの表現能力と事前学習の出来の良さには正の相関がありそうです。
(e)に注目します。 UR50/SとUR50/Dはデータセット内の多様性が大きなもので、UR100は標準化された多様性の小さなデータセットです。これを見ると同じモデルでも多様性の大きなデータセットで学習をした方がpre-trainの出来が良いことがわかりますね。
(f)ではUR50/Sのデータセットの一部だけを使って学習をしたものです。 これを見ると、データセットのサイズと事前学習の出来の良さには正の相関がありそうだとわかります。
Learning Encodes Biochemical Properties
事前学習により生化学的な特徴や表現を獲得できているか、タンパク質をembeddingし、t-SNEを用いて時限削減をしてプロットをしたのが図1。 これを見ると、疎水性・親水性、電気的特性などの特徴を捉えていることが窺えます
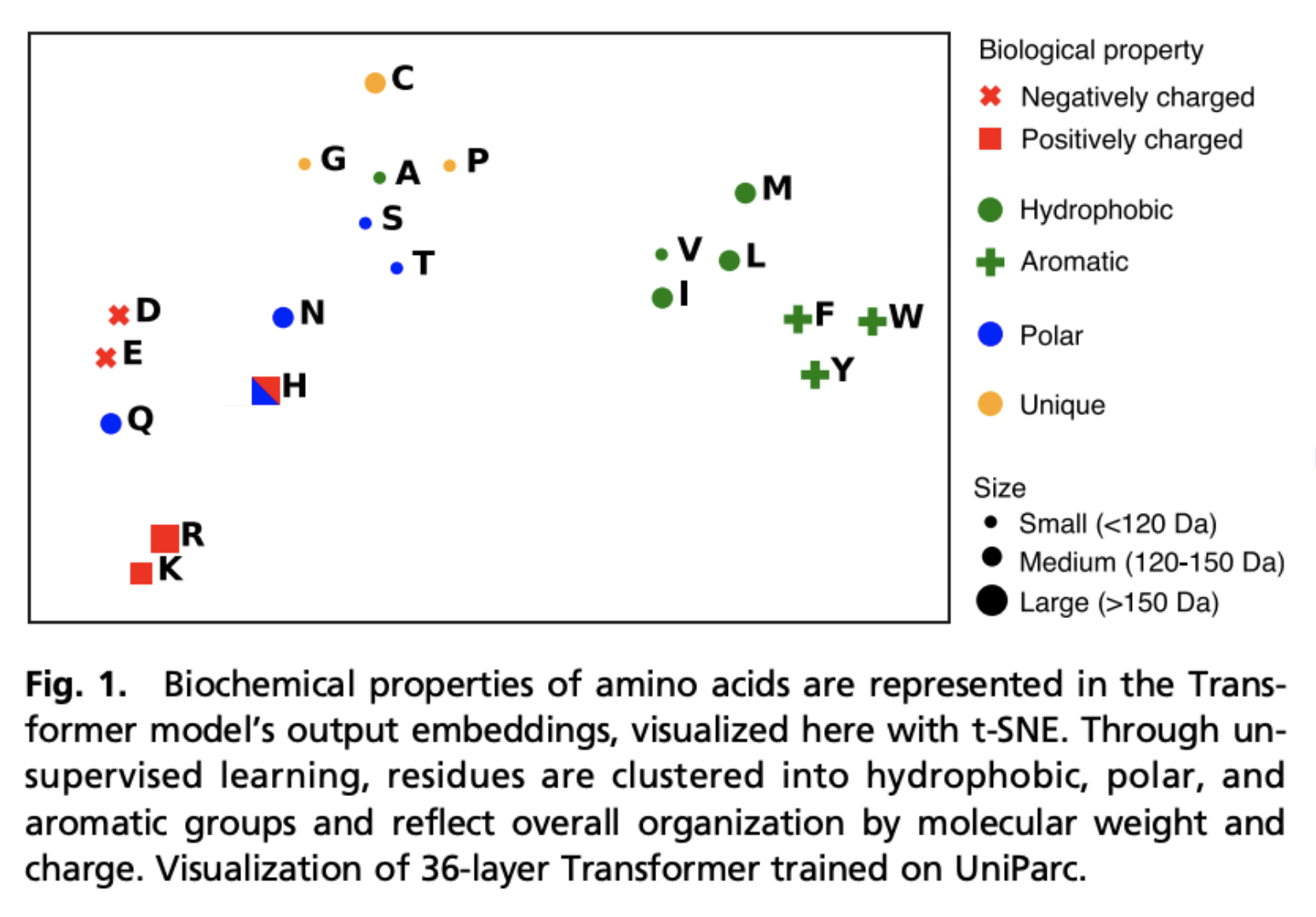
図2は学習前後の特徴空間を投影したものです。
Aを見ると、学習前では相同タンパク質は拡散している一方で、学習後にはクラスターが作られていることがわかります。
Bは線形次元削減(PCAを使用)を用いた結果を射影したもので、種による線形分離が学習により獲得できていることが予測されます。これは学習前の表現には見られないものです。

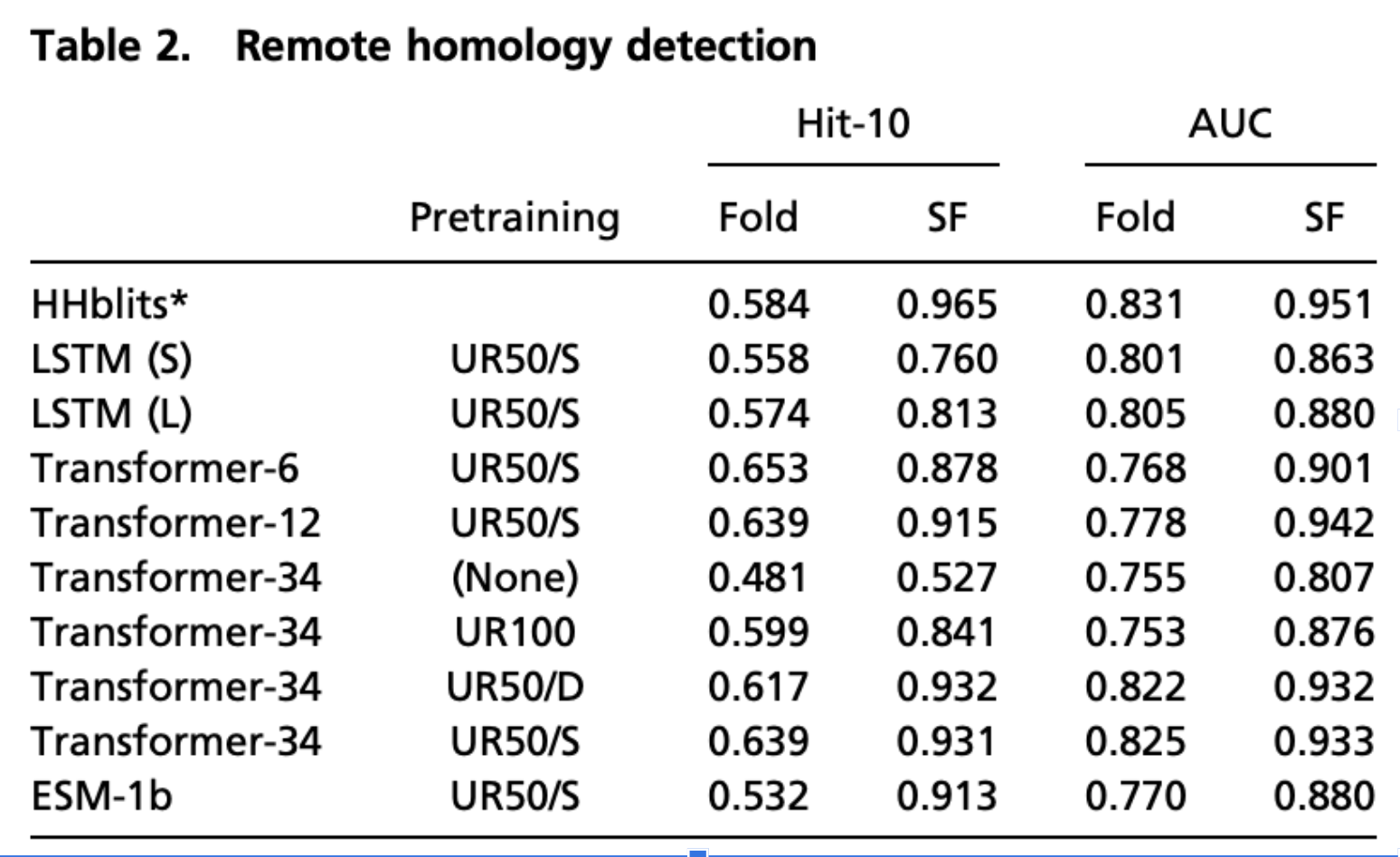
Learning Encodes Remote Homology
事前学習したモデルで相同タンパク質を分類する問題とフォールディング問題を解かせた結果が表2にまとまっている。 この分野では隠れマルコフモデルをベースにしたHHblitsという手法がSOTAである。 結果を見てみると、HHMの方が少し優秀な成績を収めている。しかし、transformerモデルも近い成績を収めている。 また、Fast vector nearest neighbor finding法は数十億の配列を検索して、クエリタンパク質との類似性をミリ秒で判定することができることもtransformerの有用性である。
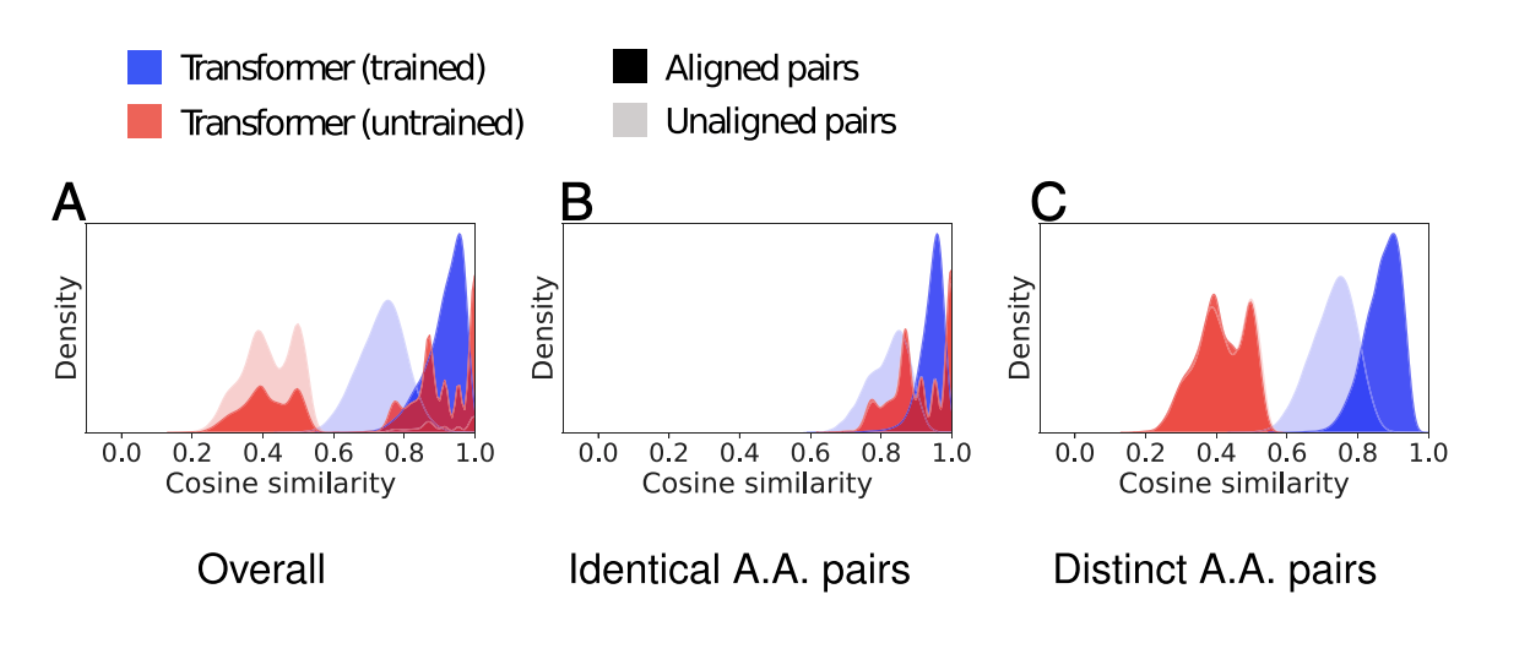
Learning Encodes Alignment within a Protein Family
図3Aは,学習したモデルとベースラインの代表的なファミリーにおける,整列した位置と整列していない位置のコサイン類似度の分布を示したものです
教師なし学習では、整列したペアと整列していないペアの分布に大きな変化が見られます
BとCをみると、このような傾向が、 同族のタンパク質ではコサイン類似度が高くなり、異なる場合が低くなるという結果を得られていることがわかりますね
Linear Projections
transformerの表現を利用して、family推定、super family推定(分子進化レベルでの類縁関係)、folding推定を5-foldの交差検証で調べた。
table3にその結果がまとめられている。 ※SSP(secondary structure projections)
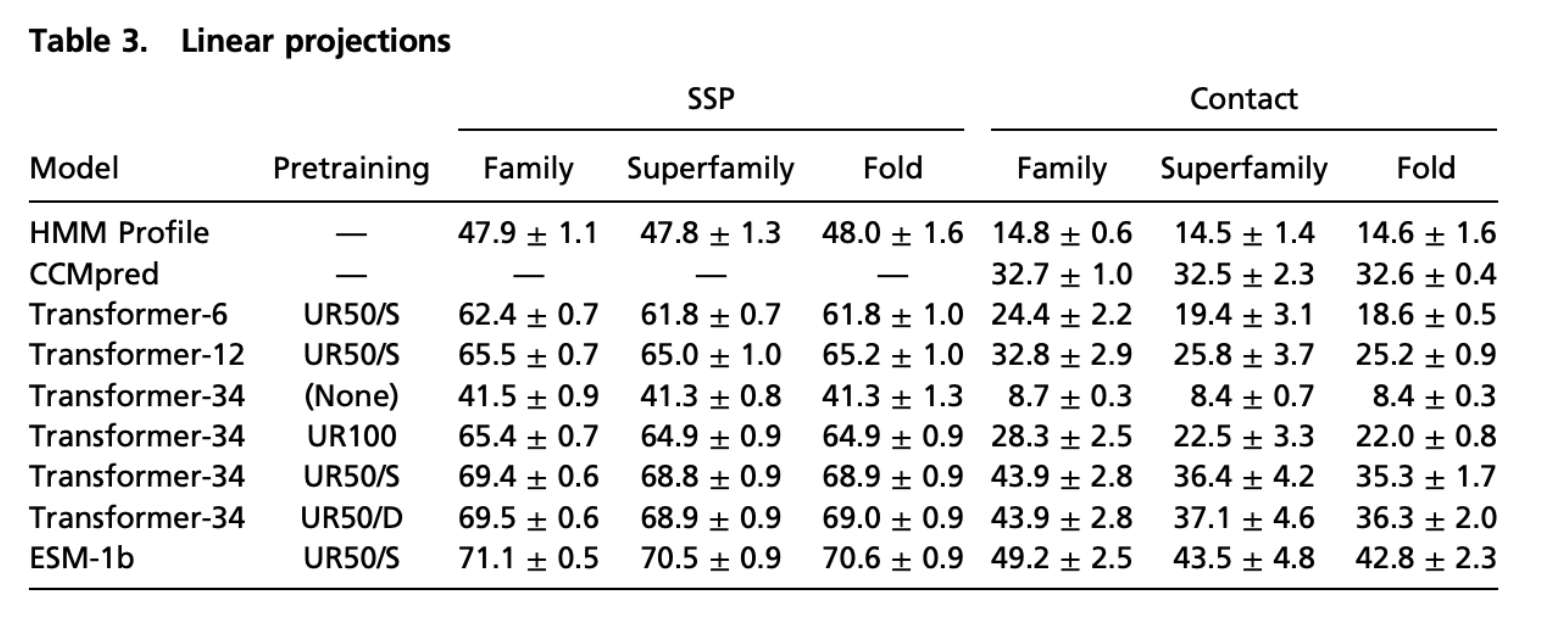
これをみると長距離のcontactについては、Transformerモデルの投影は、CCMpredによって予測された接触よりも高い精度を持っていることがわかります。
また、多様性の高い配列(UR50)を用いた学習では,二次構造と長距離連絡先の両方の学習が向上することも確認できます
Deep Neural Network
表4では二次構造の予測したスコアがまとめられています。 これをみると、LSTMよりもtransformerが優れていることが確認できます。
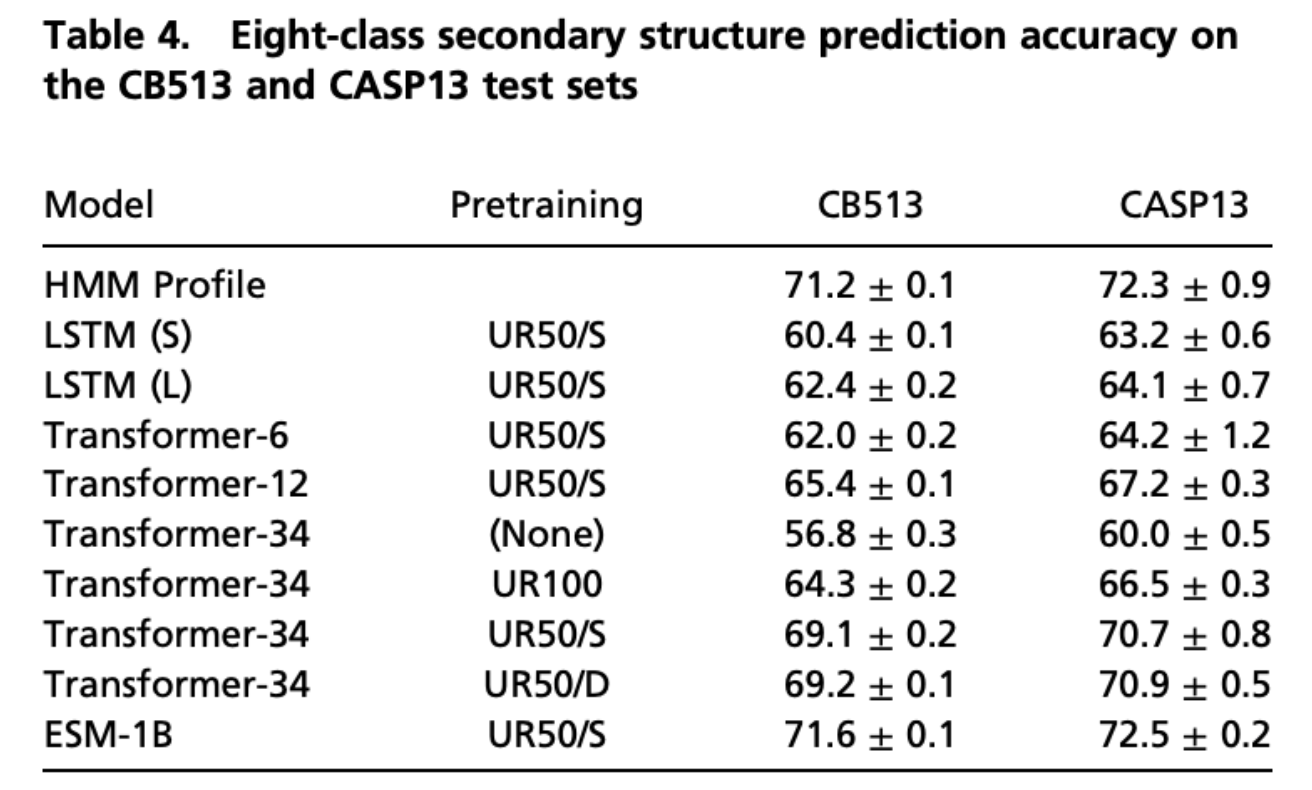
表5は長距離における接触予測に関するスコア表です
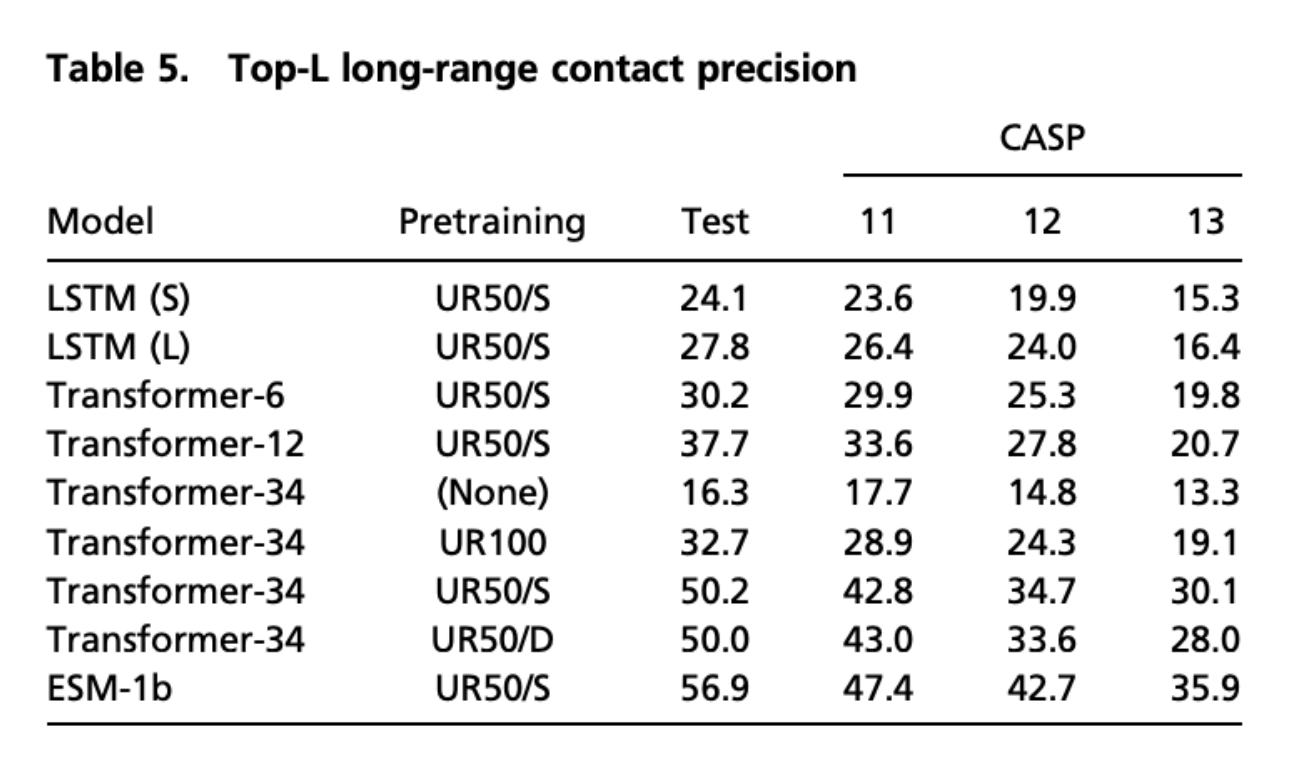
こちらに関しては、純粋なtransoformerでは50.2、ESM-1bでも56.9が最高スコアですが、従来手法のRaptorXという手法では59.4というスコアを出しているそうで、このタスクにおいてはtransformerの事前学習だけでは現状及ばずというところでしょうか。
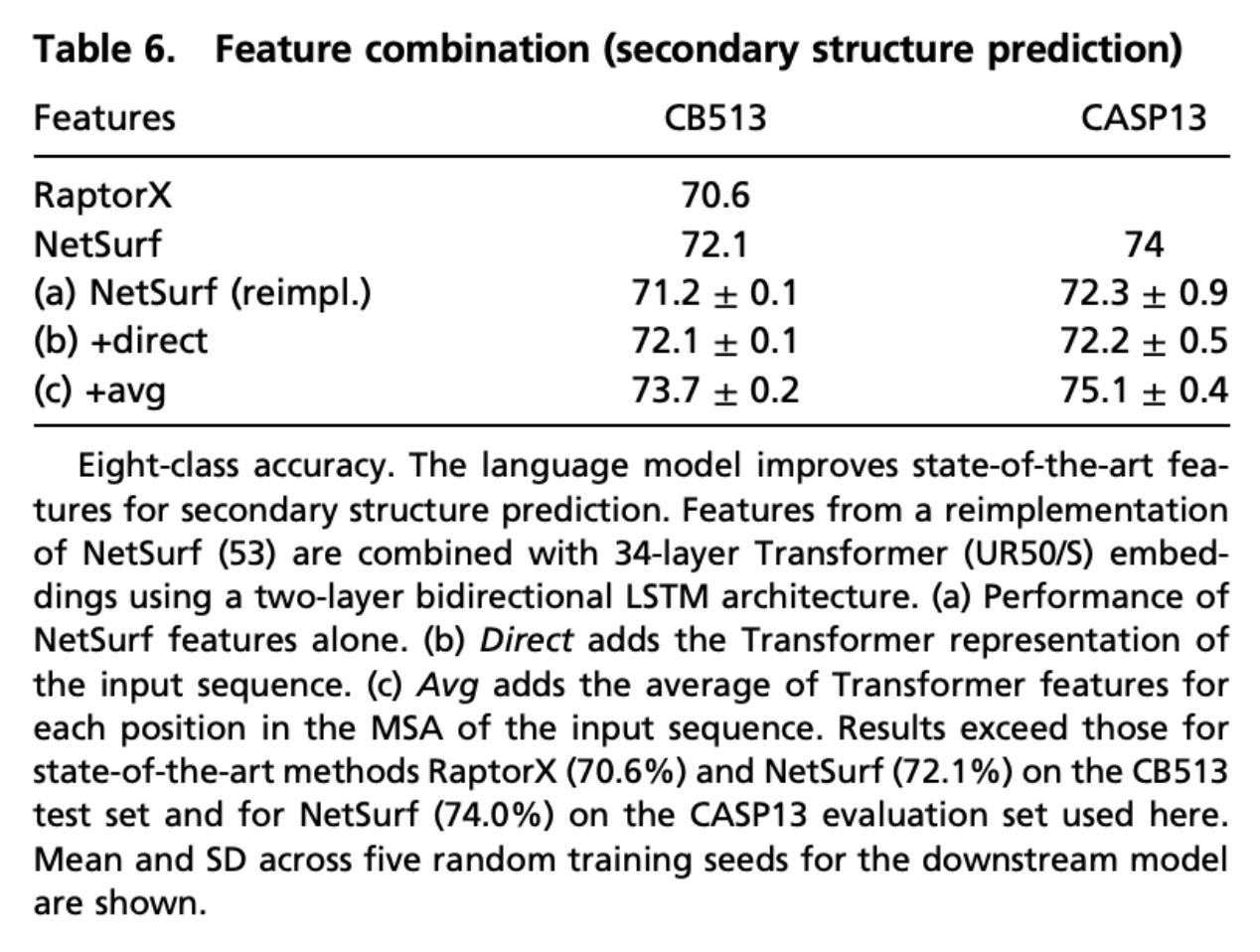
Feature Combination
教師無し学習で獲得した特徴と、SOTAを獲得している方法で取得されている特徴を組み合わせることで更なる精度改善ができるかを確認したそうです。
近年の手法では主にMSAから取得できる情報を利用することが一般的であるため、この情報をtransformerに組み込むことを考えた。
表6はMSAの情報を入力に含めた場合の比較。
b, cがコンビネーション情報を含んだスコアになっていて、比較すると入力情報にMSA情報を付加した方がスコアが高いことが確認できる。
また、pretrainしたtransformerはMSA由来の特徴にはない表現を事前学習で獲得していることも示唆している。
Discussion
論文内のディスカッションでは以下のようなことが書かれています。
生物学における人工知能の目標の1つは、ネイティブ言語(アミノ酸配列情報)で読むことができる制御可能な予測・生成モデルを作り出すこと。今後は、タンパク質の配列から本質的な生物学的特性を直接学習し、それを予測や生成に応用できる手法の研究が必要になるでしょう。私たちは、最大のタンパク質配列データベースの規模で、2億5000万個の配列から860億個のアミノ酸を対象に文脈言語モデルを学習し、調査しました。 大容量ネットワークによって配列から学習された表現空間は、アミノ酸、タンパク質、進化的相同性を含む複数のレベルの生物学的構造を反映している。二次構造や三次構造に関する情報は、ネットワークに内包されて表現されます。その際、生物の本質的な特性に関する知識は、教師なしで得られます。
経験的に、大規模なモデルによって発見された特徴は、下流のタスクでより良い性能を発揮することが分かっています。トランスフォーマーは、ベンチマークにおいて、同程度の容量を持つLSTMよりも優れています。
また、とても面白かったまとめとして
「私たちが研究しているタンパク質言語モデルは、テキスト領域で使用されているものと同程度の規模ですが、私たの実験では、まだ規模の限界に達していません。」
とも書いてありました。昨今の自然言語処理界隈ではGPT-3やswitch transformerなど数千億から数兆のパラメータを持つ大規模モデルが猛威を奮っているため、それくらいの規模のものがこの領域に入るとさらに高精度になることが期待できそうです。
そして、一番夢が広がる議論が最後にありました。
大容量の生成モデルを遺伝子合成やハイスループットの特性解析と組み合わせることで、生成的な生物学が可能になる。ニューラルネットワークが、タンパク質の配列から学んだ知識を機能的なタンパク質の設計に応用できるようになれば、予測モデルと組み合わせて、目的の機能を持つ配列を共同で生成・最適化することができるようになるだろう。
これがもし実現したら、製薬を始めとても大きなインパクトを世界中のあらゆる分野に与えることになるでしょう。
工業的に特定の機能を持ったタンパク質を合成することができるとしたら、世界がガラッと変わるかもしれませんね。
今後も目が離せません。

Ryu Ishibashi
機械学習/Vue/React/Laravelとかやってます